Renklerin İfade Etmek İstedikleri
Veri görselleştirme, verinin kendisi kadar önemli bir alan. Bilgiyi alıcıya aktaracak olan bu görseller. Kaldı ki bizler gibi büyük veri (big data & meta data) ve birçok disiplini bir araya toplayan disiplinlerarası (interdisciplinary) çağında yaşayan bilim insanları için, “araştırma hikayesini” anlatabilmenin en önemli yollarından biri de bu görsel sunum şölenleri olan veri görselleştirmeler.
Kişisel olarak da bilimin en renkli ve eğlenceli hallerinden biri olarak tanımlıyorum. Her gördüğüm yeni plot türünü deneme fırsatı yakalacağım o anı bekliyorum.
Öte yandan, o kadar çok plot türü var ki insan hangisini hangi veri için seçeceği konusunda zorlanıyor. Bu konuda grubumuzun aktif üyelerinden Melike Dönertaş’ın yapmış olduğu sunumu izlemenizi tavsiye ederim.
En bilinen RNA dizileme amacı olan diferansiyel gen ifadesi analizi ile başladığımızı düşünelim. RNA’yı izole ettik, sekansladık, sonra referans bir genomla eşleştirdik (mapping–alignment), hangi genin ne kadar ifade edildiğine dair sayıları elde ettik (count) ve sonra farklılaşmış gen ifadesini (DEGs) analiz ettik (örneğin Deseq2 ya da egdeR kullanarak). Sonra da bunu görselleştirmek istedik.
Volkan (Yanardağ) Plot
Karşılaştırılacak iki durum olduğunu varsayalım (ilaç uygulanmış vs. kontrol). İlaç uygulandığında bazı genlerin ifadesi değişmiş-Differentially Expressed Genes, DEGs– (normalde fazla (upregulated) ya da az (downregulated) ifade olmuş olsun, bazıları da aynı kalmış, etkilenmemiş) olsun.
Artan/azalan gen ifadesi ve bunun p-değerlerini bir grafikte göstermek istediniz. Bazı genler yüksek/düşük ifade oluyor gibi gözükse de p-değeri yüksekse bu farkın bir önemi yok.
Soru: İstatistiksel olarak önemli kaç/hangi gen fark yarattı? Herhangi bir trend var mı?
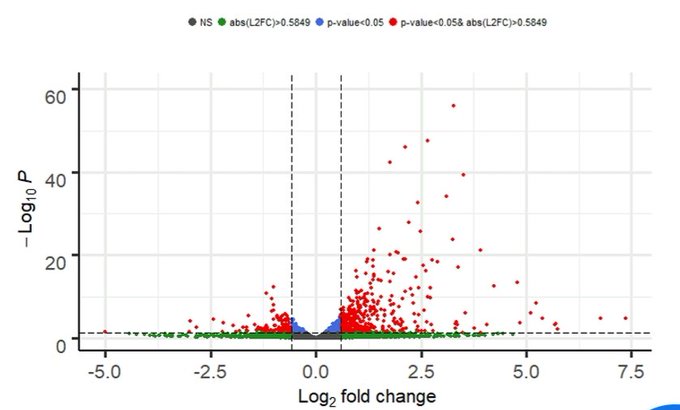
Genelde p değeri -log10 tabanında gösterilir (yani p<0.05 değeri 1 ile 2 arasında bir skalaya gelir-y ekseninde). P değeri 0.05’ten küçük olan her değer, y ekseninde daha büyük değere tekabül edecek.
Genelde DEG ifadesi de log2 tabanında gösterilir (yani mutlak log2FC, fold change (kat değişimi), değeri 0.5849 aslında 1.5 katı demek. Bu da birinde mesela 15 olan ifade, diğerinde 10 demek). Artan genler sağda, ifadesi azalanlar da solda kalıyor x ekseninde.
NS: Not significant (ne log2FC değeri sınırı aşabilmiş ne de p-değeri), abs: absolute (mutlak değer, artı eksisi olmadan) Griler, artıp azalma sınırının altında kalmış. Mavilerin değişimi önemli (p-değeri) ama değişim miktarı (log2FC) sınırın altında. Bunlar şu an ilgimizi çekmiyor.
Yeşillerin değişim miktarı (artış/azalış), sınır olan log2fc:0.5849’u aşmış ama p-değeri=>0.05 olduğu için sınıfta kalmışlar. Kırmızılar da hem değişim miktarı (ifadede artma azalma) büyük, sınırın üstünde, hem de karşılık gelen p-değeri rastgele olamayacak kadar önemli (0.05<). Kırmızılar, iki açıdan da (hem log2FC hem de pvalue) sınır değerini (treshold) geçen istatistiki olarak önemli değişime sahip azalan (x ekseninin solunda) ve artan (x ekseninin sağında) genleri ifade ediyor.
İlgili Kod
#İlgili kodun orjinali için şu siteye gidebilirsiniz: https://www.bioconductor.org/packages/release/bioc/vignettes/EnhancedVolcano/inst/doc/EnhancedVolcano.html
makeVolcanoPlot = function(df, mutant = 'AChE mutant', pCutoff = 0.05, FCcutoff = 0.5849) {
EnhancedVolcano(df,
lab = row.names(df),
# eğer aradığınız bazı genleri nerde olduğunu görmek istiyorsanız, dosyanızda hangi isimle eklediyseniz, ensembl id, gene name, vs. onları select lab ile seçerek grafikte yazılı olarak görmek mümkün.
#selectLab = c("arr3a","ache","fabp10a","pck1","rpe65a",
# "mylz3","desma","rom1b","sagb","slc4a5"),
x = 'log2FoldChange',
y = 'pvalue',
# başlık eklemek isterseniz, title ı kullanabilirsiniz
title = paste(mutant, ' vs. Healthy'),
#subtitle ile ek alt başlık eklemek de mümkün
subtitle ="",
caption = paste0('Total = ', nrow(df), ' genes'),
captionLabSize = 10,
titleLabSize = 16,
subtitleLabSize = 1,
axisLabSize = 14,
transcriptPointSize = 1.0,
transcriptLabSize = 3.0,
# boxedlabels = TRUE,
pCutoff = pCutoff, #horizontal cut off line
FCcutoff = FCcutoff, #vertical cut off line
legend=c('NS',paste('abs(L2FC) > ', FCcutoff ),
paste('p-value < ', pCutoff),
paste('p-value<', pCutoff, '& abs(L2FC) > ', FCcutoff)),
legendPosition = 'top',
legendLabSize = 10,
legendIconSize = 3.0,
colAlpha = 1)
}
Peki ya daha fazlası mümkün mü?
Aslında bu grafik size, kırmızıları diğerlerinden ayırt etmekten biraz fazlasını sunuyor. Belki işte bu sebeple, veri görselleştirme ile yepyeni ufuklar kazanabilir, yepyeni sorular üretebilir ve yepyeni bir bakış açısı kazanabilirsiniz.
Bizim yukarıdaki grafik üzerinden anlatmak gerekirse,
- artan ifadesi olan (x ekseni sağ taraf) gen sayısının azalış gösterenlere göre fazla olduğunu (x ekseni sol taraf)
- artan ifadesi olan genlerin pek çoğunun önemli değişim değerine sahip olduğunu (x eksenindeki kırmızılar)
- özellikle log2FC, kat değişimi, 0.5849 ile 2.5 arasında olan genlerin (x ekseninde sağa ve sola doğru nokta nokta dikey çizgiler ile 0 noktasında kalan alan), değişim miktarının verilen veri seti içinde uyumlu bir şekilde artış gösterdiği, rastgele olmadığını (önemli olarak kendini gösterdiğini)
- log2FC değeri büyük değişim gösteren genlerin p değerinin de o derece düşük olduğunu (y ekseninde artan değer, aralıklı dağılmış kırmızı noktalar)
- artış gösteren gen ifadesindeki değişimlerin azalış gösterenlere göre daha büyük olabileceğini (x ekseninin sağ tarafının sol tarafına göre daha uzun devam eden çizgide hala noktalara sahip olması) ve bunların önemli değişime sahip olması (kırmızı nokta olması) tek bir grafikten okuyabiliyoruz.
Bir arkadaşımın sorusu ile başlayan volkan plot grafik yorumlama serüvenimiz diğer plotlar ile devam edecek. Umarım sizler için de faydalı olmuştur. Böyle düşünüyorsanız, sizler de bu yazıyı paylaşarak, büyük paylaşımcı ailemize destek olabilirsiniz.
Sağlıcakla kalınız.
