Covid19 ile hiç duymamış olanların bile öğrendiği bir lab tekniği olan, moleküler bioyolojide yeni bir devir açan Nobelli teknik PCR (Polymerase Chain Reaction)’dan esinlenen bir method olarak kullandığımız qPCR (quantitative PCR)’ın temelinde iyi bir primer dizaynının yattığını söylemek yanlış olmaz sanırım. Kısaca, hedeflediğiniz sekansı çoğaltmak için baştan ve sondan 15-25 nucleotid dizisi uzunluğundaki “primer”leri polimeraz enzimi için başlangıç dizisi olarak kullanıyoruz.
Bir labda çalışmaya başladıktan sonra belki ilk biyoenformatik deneyiminizi gerçekleştirmenizi sağlayan NCBI-BLAST’tır. Peki, bu yoğun bilgi taşıyan siteden nasıl amaçladığımız bilgi ile çıkacağız?
Lablarda sıkça kullandığımız qPCR gibi teknikler için primer dizaynına dair bazı püf noktaları sizlerle paylaşacağım.
NCBI ile hedef genimizle ilgili detaylara ulaşma
NCBI ana sayfaya gittkten sonra “Search” (Arama) kutucuğuna genimizin ismini yazarak (ACTB genini örnek seçtim), “Gene” sekmesi altında aratıyoruz.
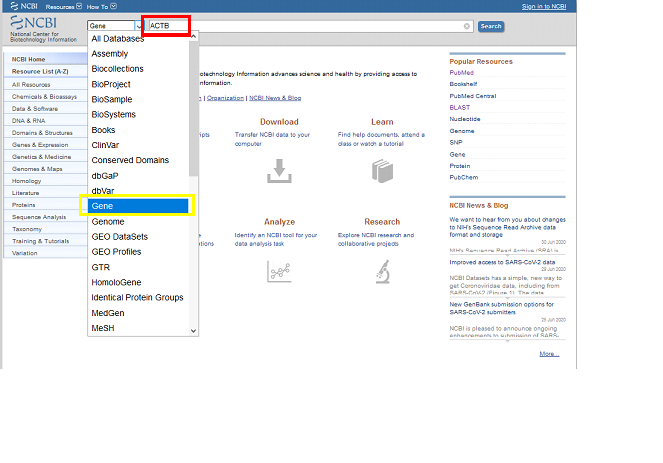
Daha sonra karşımıza bir süre seçenek çıkıyor. Bunlar aslında diğer canlılarda insandaki bu genin karşılığı olanlar (ortholog) ya da varsa eski bir zaman diliminde ikilenmiş (duplication) ve belki sadece sekans değil işlev (function) olarak da farklılaşmış insandaki benzer genler (paralog)a sizleri yönlendiren bir sayfa bu (neyden bahsediliyor diyenler için link).
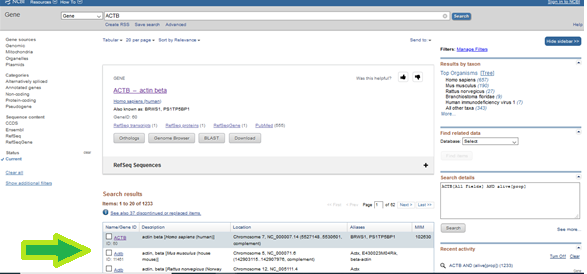
Burada ilgili genle ilgili pek çok bilgiye erişmeniz mümkün. Exonic intronic bölgeler, genin bulunduğu kromozom, tam lokasyonu, Expression kısmında hangi dokularda ifade olduğunu, veyahut hangi fenotiple ilişki içinde olduğunu, tek nüklotit varyantlarını (SNPs), vs. pek çok detayını öğrenmek mümkün (bu noktada bir diğer alternatif UCSC Genome Browser olacaktır).
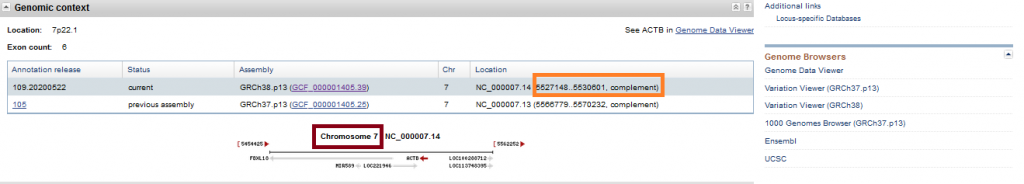
Biraz daha aşağı inerseniz, bu genin tüm transcriptlerini (buradan ifade olunan RNAlar) görebilirsiniz.
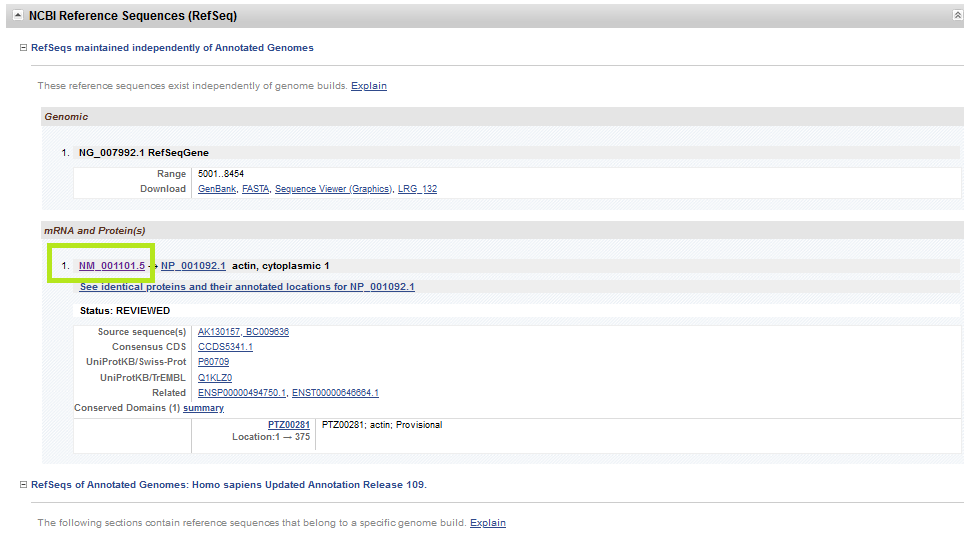
Şimdi ilgili genle ilgili mRNAnın detaylarına aslında NCBI-Gene yerine NCBI-Nucleotide kısmından da erişilebilir olduğunu göreceksiniz.
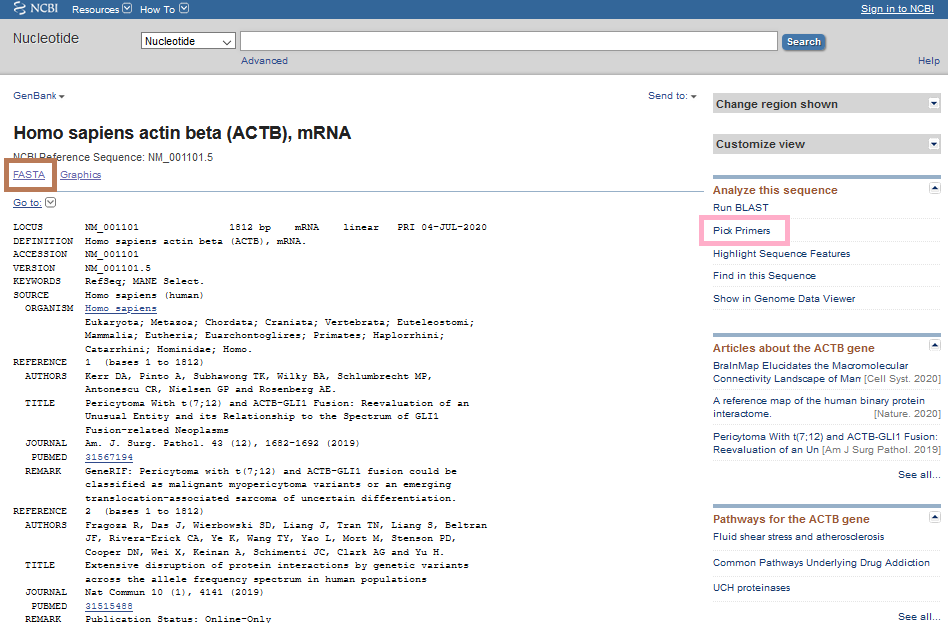
Burada önümüze iki seçenek çıkıyor. Birinci sağdaki pembe kutu ile gösterilen Pick Primers seçeneği ile Primer-BLAST‘a yönlenip oradan primer dizayn etmek, ikincisi ise sol üst köşedeki kahverengi kutudaki FASTA seçeneğine tıklayıp, ATCG harflerinden oluşan sekansı kopyala yapıştır yaparak başka primer dizayn araçlarını da denemek (mesela Primer3). Her ikisini de deneyeceğiz.
Primer-BLAST
Pick Primer dedikten sonra sizi şöyle bir sayfaya yönlendirecek:

Buradan sonra primerinizle ilgili bazı değerleri değiştirebiliriniz. Sağ yanlarındaki soru işaretine tıklayarak, her bölmenin hangi değişken için olduğunu detaylı bir şekilde öğrenebilirsiniz. Biz şimdi kısaca birkaç tanesini ele alacağız.
PCR product size ile tam olarak pcrda çoğaltmak istediğiniz hedefin uzunluğunu kısıtlayabilirsiniz. Bu noktada, qPCR’da kullandığımız SYBR-Green vs.nin en fazla 500bp hedef uzunluğu olmasına rağmen, 100-200 arası bir hedef ile optimumu olduğunu unutmamanızı öneririm.
Turuncu karedeki özellikle, aradığınız özelliklere uygun primer sayısını sınırlayabilirsiniz. Mesela, bana sadece ilk 10 tanesini göster gibi.
Mavi karedeki özellikle de primerinizin erime sıcaklığı /Tm) özelliklerini seçebilirsiniz. Tm en düşük 57, en fazla 63 olan ve aralarında en fazla 3 derece erime sıcaklığı farkı bulunan primerleri seçmek istiyorum gibi.
Yeşil karedeki özellikle de aradığınız primerin rastgele bir gen bölgesini mi yoksa exon-exon birleşimini hedef seçmek istediğinizi seçebiliyorsunuz. Amacınıza göre değişmekle beraber, eğer hiç bilmeden bu işe giriyorsanız, direk exon-exon tarayan seçeneğini seçmenizi öneririm (detaylı bilgi).
Kahverengi karedeki “submit” seçeneği ile ne olacağını görebilirsiniz. Burada “show results in a separate window” ile ayrı bir pencerede işleminizi gerçekleştirip, değişiklik için parametre değiştirme penceresini olduğu yerde bırakabilirsiniz.
Eğer ki daha fazla parametre ile ilgili bilgi sahibi iseniz, submit butonu aşağısındaki “advanced parameters” seçeneği ile diğer seçenekleri de düzenlemek mümkün.
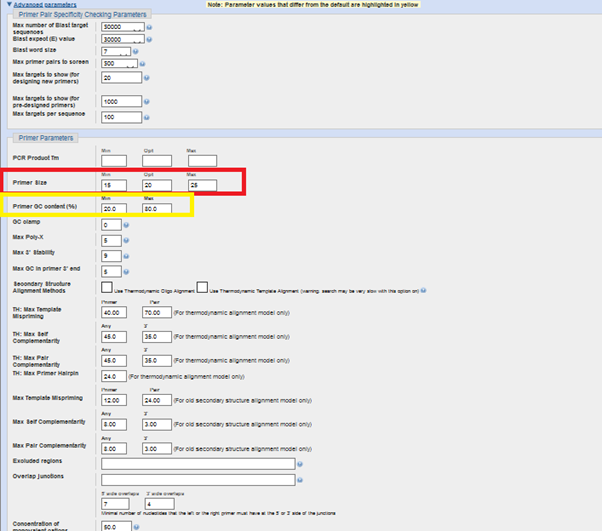
İleri parametrelerde de özellikle “primer size”(primer uzunluğu) ve primer sekansı içerisindeki GC miktarı (yüzde olarak) (“Primer GC content), size göre optimize etmek mümkün. (Ben bu noktada GC için 40-60, primer çifti uzunluğu için de 18-20-23 seçeneği tercih ettim. GC-AT farklılığı bağ sayısına bağlı olarak primerlerin verimliliğini değiştirebilir.)
Submit ettikten sonra, şu şekilde biraz bekleyeceğiz.
Evet, daha sonra çıkan sayfada, primerinizin tam olarak nereyi çoğaltacağını ve diğer özelliklerini (bağlanma sıcaklığı, kendi kendine birleşiyor mu, diğer primerle birleşiyor mu vs. gibi) bulacaksınız.
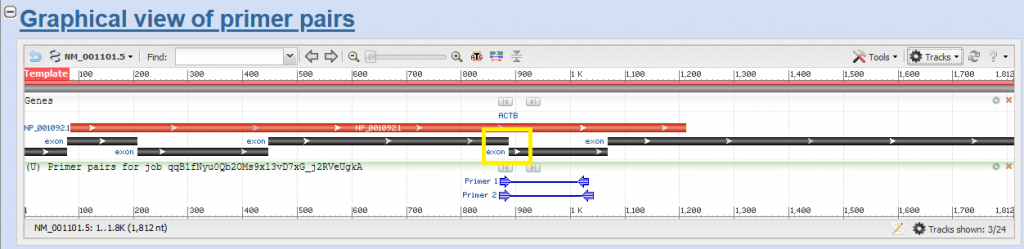
Siyah kutucuklar, adı üstünde exonlar. Sarı, içi boş kutucuk da exon-exon bölgesini tarayan bölgelerden birini gösteriyor. Kırmızı kısım, protein-kodlayan kısmını. Aşağısındaki iki küçük mavi çizgi de primer dizayn aracının verdiğimiz kritere göre bulduğu iki çeşit primer çiftinin çoğalttığı hedef bölgesini. Öte yandan, transcriptin sonunu hedeflemek istemiyoruz. Çünkü rnalar bozunuma uğruyor (degredation). Bu da özellikle 3′ (sağ uç) sonu etkiliyor. 5′ (sol uç) ise bi tık daha güvenli. O yüzden primerlerimiz biraz daha sol uca yakın olsa daha iyi olabilirdi.
Primer çiftlerimizin ikisi de şimdilik kriterlere uygun gözükse de bakalım gerçekten aradığımız gibi mi?
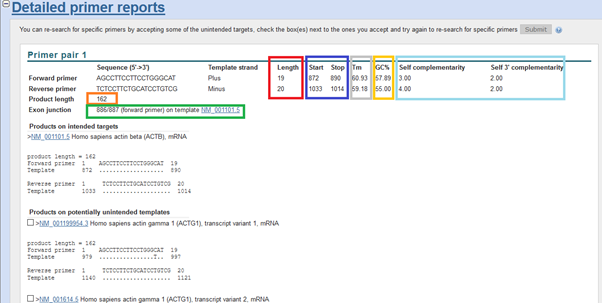
Primer çifti raporun baktığımızda görüyoruz ki primerin çoğaltacağı hedef uzunluğu 162 ile ideal sınırlar içerisinde (100-200). Exon-exon birleşim bölgesinde. Primer uzunlukları da ideal: 19 ve 20. Tm de 60<= civarı. GC içeriği de (çünkü AT, GC oranı benzer olmasa bu da bağlanmadan kaynaklı farklılıklar oluşturabilir) 40-60% aralığında. Biraz kendi kendine (self-complementarity), bağlanma ihtimali var (istenmeyen primer dimers), ama o da kabul edilebilir oranda diyebiliriz. Ancak, aşağıda tüm varyantlar dahil diğer olası hedefleri gösteren (mesela başka bir transcripti hedef alıyorsa, actb yerine, bu sefer çıkan sonuçtaki miktarın o gene ait olup olmaycağını bimediğimiz için sıkıntılı bir durum.) bir liste var primer çiftleri için.
Ve maalesef hedef dışı transcriptler görüyoruz, bu primer çiftleri ile çoğalabilecek. Bunu, in silico PCR ile doğrularsak eğer, bu durumda bu primer çiftleri, maalesef uygun olmayacak. Yeniden dizayn etmemiz gerekecek. Bu durumda ya kendiniz dizayn edebilirsiniz ki bu aşırı yorucu bir süreç, ya da alternatif diğer araçları (benim favorim primer3) kullanabilirsiniz.
Primer3
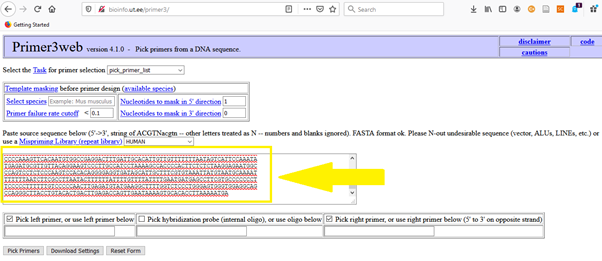
Sizi primer3′de şöyle bir sayfa karşılayacak, burada sarı okla gösterdiğim bölgeye NCBI’dan kopyala yapıştır ile mRNA FASTA sekansı yapıştırın:
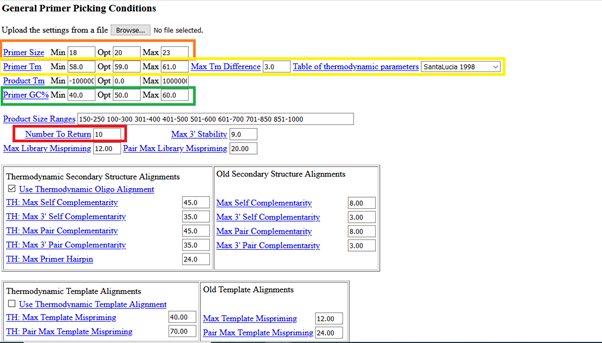
Ve tabiki burada da primer uzunluğu (primer size), hedef uzunluğu (target size), GC %, minimum-maximum Tm değerleri gibi değiştirebileceğiniz faktörler var. Biraz aşağı inerseniz aynı sayfada, şunları görebilirsiniz:
Seçimlerinizi yaptıktan sonra “pick primers” e basarsanız, şöyle bir sonuç alacaksınız:
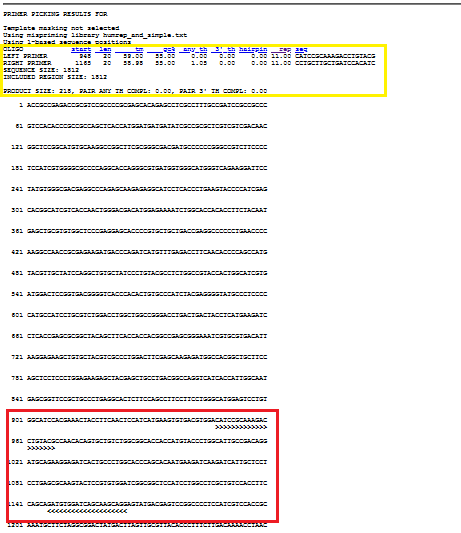
Sarı kutuda ilk primer çitfinin detayları ve kırmızı kutuda, hangi bölgesini hedeflediğini gösteriyor.
Aşağı indiğiniz takdirde ise alternatif 9 aday primer çifti daha görebiliyorsunuz.
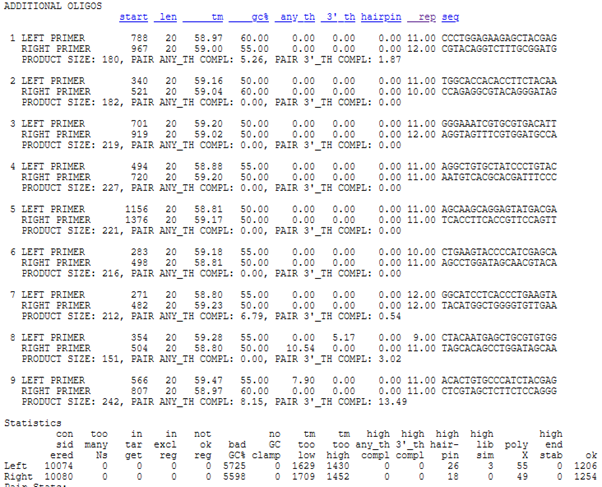
Daha önce sol uca yakın seçmek istediğim için, 100-200 aralığındaki 2. alternatif dikkatimi çekti (ancak 1. ve 8.yi de alternatif olarak deneyeceğim eğer 2de arzuladığım sonucu alamazsam). In silico PCR ile tam olarak doğru olup olmadığını tespit edeceğim. (Bu arada diğer parametre seçenekleriyle ilgili bilgilere, mavi renkli başlıklarına tıklayarak ulaşabilirsiniz.)
In silico PCR
qPCR yapıp bir sürü materyali kullanmadan önce, programların dizayn ettiği primer çiftlerinin gerçekten hedefimizi mi yoksa başka bir şeyleri mi çoğalttığını görmek istiyoruz.

Öte yandan exon-exon bağlantısı tarıyor seçeneğini primer3’de seçememiştik. Burada konfirme edeceğiz. Eğer arada en az bir intron varsa, ve yaptığımız PCRdaki hedeften daha uzun bir sonuç elde etti isek (agarose jelde yürüterek tespit edebilirsiniz), bunun sebebinin bir dna kontaminasyonu olduğunu tahmin edebiliriz (teoride transcriptte intron beklemiyoruz). Kısacası, PCR tüm çabalara rağmen çalışmadığı takdirde, bunun sebebine dair bize ipucu sunabilir. Öte yandan, eğer ki başka hedefleri de çoğaltıyorsa (multiple targets), bu durumda bunu UCSC’un in silico pcr ile dry-lab (bilgisayar üzerinden) görmek mümkün.
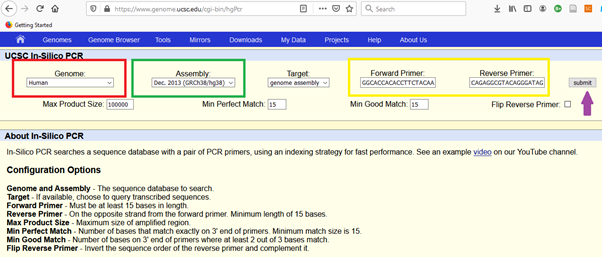
Hangi canlı genomundasa ilgili transcript, onu seçiyoruz. Sonra en son (latest) “assembly” yi seçiyoruz. Seçilen baş ve son uç primer çiftimizi kopyala yapıştır yapıyoruz.
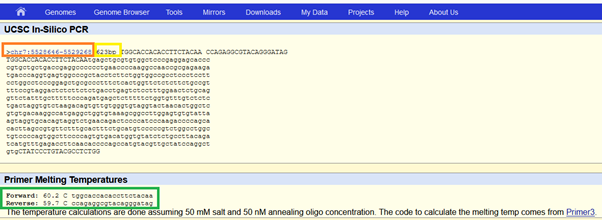
In silico PCR sonucunda gördük ki hedefimiz 7. kromozomda, ACTB genimizin bulunduğu lokasyonda bir yerleri çoğaltıyor. Ve tek hedefi var. Üstelik, arada en az bir intron var (exon-exon taradığına emin olduk), çünkü qPCR hedefimizin gittiği gende taradığı bölge hedefin kendisinden uzun (182 vs. 632).
Referans Gen
qPCR’ı gen ifadesini ölçmek için yaptık. Öte yandan, hedef genlerimizin yanı sıra, örneklerdeki gen ifadelerindeki farklılığın gerçekten bizim değiştirdiğimiz durumdan ötürü (mutasyon, bir ilaç vs. olabilir bu) mü yoksa aslında bazı teknik hatalardan (örnekleri eşit miktarda koymamak vs.) mı kaynaklandığını görmek için referans gen/mRNA adı verilen (reference or housekeeping) başka genleri/mRNAları de kontrol ederiz.
Burada dikkat edilmesi gereken unsur, bu seçilen referansın, her dokuda ve her durumda, etkilenmeden aynı derecelerde ifade olmasıdır.
Bu sebeple eskiden beri Actin ve GAPDH gibi genler referans seçilirdi. Ancak son yıllardaki çalışmalar gösterdi ki bu genler de o kadar sabit bir ifade profili sergilemiyor. Bu yüzden şimdilerde çalıştığınız durum için (çeşitli kanser türü ya da yaşlanma olabilir) en uygun referansı seçmek için literatürü sıkı bir takip öneririm.
Kanser türleri için tek tek referans gen için bir liste barındıran şu makale ile:
Conventionally used reference genes are not outstanding for normalization of gene expression in human cancer research (burada önerilenler: • HNRNPL • PCBP1 • RER1)
bu konuda diğer ufuk açıcı bilgiler sunan bu makaleyi tavsiye ederim:
Human housekeeping genes, revisited
Kısa kısa qPCR sonuç hesaplama
(Daha önce hazırlamış olduğum örnek taslağı sizle de paylaşıyorum.)
qPCR sonuçlarını analiz ederken neyi görmek istiyoruz? Göreceli ifade edilmeyi (yani, A geni, B genine göre 10 kat artmış vs gibi) mi yoksa mutlak değerleri mi (A geni, şu kadar ifade edilmiş, B geni de bu kadar vs. gibi)? Öncelikle bunun ayrımını yapıyoruz. Göreceli ifade edilmede de yine kontrol genleri kullanıyoruz ki her bir geni birbiriyle kıyaslayabilelim.
qPCR ile veri analizinde, bir 2^(-𝚫CT), 2 üzeri delta Ct demek, methodu var bir de 2^(-𝚫𝚫CT), 2 üzeri delta delta Ct diye okunur, methodu var.
| Örnekler | Referans gen ve baktığımız hedef gen | |
| CTb-actin (reference/housekeeping) | CTgtg1l3 | |
| kontrol (sağlıklı) | 18.0 | 17.0 |
| deney (hastalıklı) | 19.0 | 14..0 |
- method [2^(-𝚫𝚫CT)]
İlk önce hedef genimizi/gbtg1l3) referansa(b-actin) göre normalize ediyoruz. Daha sonra hasta Ct değerini sağlıklıya göre normalize ediyoruz. Sonra da ifade edilme oranını buluyoruz (fold-difference):
𝚫CT (sağlıklı) = 𝚫CT(gbgt1l3) – 𝚫CT(b-actin)
𝚫CT (sağlıklı) = 17-18 = -1.0
𝚫CT (hasta) = 𝚫CT(gbgt1l3) -𝚫CT (b-actin)
𝚫CT (hasta) = 14-19 = -5
𝚫𝚫CT= 𝚫CT(hasta) -𝚫CT(sağlıklı)
𝚫𝚫CT= -5 – (-1) = -4
2^(-𝚫𝚫CT)= Normalize edilmiş ifade = 2^(-4) = 16
Bu ne demek? Hastamızda gbgt1l3 geni 16 kat daha fazla ifade edilmiş (sağlıklıya göre).
2. method [2^(-𝚫CT)]
Burada da ilk önce her bir örnek için(hasta vs. sağlıklı) ilgili genlerin (gbgt1l3 vs. bactin) göreceli ifade edilmesini hesaplıyoruz. Şöyle:
2^(𝚫CT of bactin – 𝚫CT of gbgt1l3) = Göreceli ifadeyi burdan buluyoruz.
Sağlıklı için = 2^(𝚫CT of bactin – 𝚫CT of gbgt1l3) = 2^(18-17) =21=2
Hasta için = 2^(𝚫CT of bactin – 𝚫CT of gbgt1l3) =2^( 19-14) =25= 32
Sonra da bu ifadeye karşılık gelen ifade oranını hesaplıyoruz. Şöyle:
Sağlıklının ifadesi = sağlıklı/sağlıklı =2/2=1
Hastanın ifadesi = Hasta / sağlıklı = 32/2=16 kat artan bir gbgt1l3 ifadesi var.
Ekler
Dokularda gen ifadesi
Bu arada geniniz hangi dokularda daha çok ifade oluyor, hangi hastalıklarla ilgili vs. gibi bilgi edinmek isterseniz, kısa kısa GeneCards ve ExpressionAtlas‘tan istifade edebilirsiniz.
Primer Verim Hesabı
Bunlardan ayrı olarak bir de verimi hesaba katarak yaptığımız ölçümler var. Verimden kastım nedir? Bu primerler nasıl çalışıyor? Yani PCR’da ilgili genle eşleşen bu primerler, her cycle (döngü)da gerçekten iki katına çıkarıyor mu genimizi bilmiyoruz. Yukarıdaki metodlarda, iki katına çıkararak güzelce çalıştığını/ya da gbgt1l3 ve bactin primerlerinin aynı verimde çalıştığını varsaymış olduk. Ama öyle mi?Aslında her primeri, ne kadar iyi çalıştığı konusunda test etmemiz lazım. Bunu da elimizdeki primerleri değişik miktarlarda dilüe edilmiş cDNA’larla deneyerek (serial dilution) hesaplayabiliyoruz.
Neden dilüe ediyoruz? Şöyle ki eğer ben bu primerlerin karışımdaki oranını yarıya indirirsem ne olur? Benim primerlerimin miktarı da yarı yarıya düşecektir. Eğer ki 4 katı düşürürsem ne olur? Her seferinde ilgili karışım, treshold/sınır (Ct) değerine daha geç ulaşacak demektir. Ya da bunun tam tersini (artırmak) düşünebilirsiniz.
Sonra çıkan değerleri GraphPad/excel’e koyacağım ve yatay bir eğri/çizgi elde edeceğim. Bu ne demek? Böyle güzel bir çizgi, primerlerimin güzelce çalıştığının bir kanıtı.
R: correlation yani ilişkiyi gösterir. Burada ilişki değerinin +/-1’e yakın olması, primerlerin ilgili geni her döngüde (cycle) 2’ye katladığının kanıtıdır.
Neden log?
Çünkü
log22 =1
log24=2
log21/2= -1
log21/4=-2
….
Yani, ikiye katlandığında bir fold-change alırsınız. Yarıya düşürdüğünüzde -1 fold-change olur. 2 katına çıktığınızda 2 fold-change olur.
Ya da seri dilüsyonda uyguladığınız metoda göre log10’i de kullanabilirsiniz.
Kısacası, katlanma:/azalma vs. gibi durumlar için belli bir aralıkta, düzgün (straight) bir ilişki (correlation/R) çizgisi elde edebilirsiniz.
Teşekkür
Yüksek lisansım boyunca üyesi olduğum ve bana her açıdan katkı sağlayan, değerli hocam Özlen Konu ve Konu Lab‘da en basit işlerden en komplex analizlere kadar bende emeği geçen çok kıymetli dostlarıma (özellikle Ayşe G. Keşküş, Said ve Seniye Targen) teşekkürlerimi sunarım.
References
- Nobeli PCR: https://www.nobelprize.org/prizes/chemistry/1993/mullis/lecture/
- NCBI websitesi: https://www.ncbi.nlm.nih.gov/
- Ortholog/paralog: Jensen, R.A. Orthologs and paralogs – we need to get it right. Genome Biol 2, interactions1002.1 (2001). https://doi-org/10.1186/gb-2001-2-8-interactions1002
- Exon-exon junction: https://sg.idtdna.com/pages/education/decoded/article/use-splice-junctions-to-your-advantage-in-qpcr
- Primer3: http://bioinfo.ut.ee/primer3/
- UCSC: https://www.genome.ucsc.edu/index.html
- In silico PCR: https://www.genome.ucsc.edu/cgi-bin/hgPcr
- Eisenberg E, Levanon EY. Human housekeeping genes, revisited [published correction appears in Trends Genet. 2014 Mar;30(3):119-20]. Trends Genet. 2013;29(10):569-574. doi:10.1016/j.tig.2013.05.010
- Jo, J., Choi, S., Oh, J. et al. Conventionally used reference genes are not outstanding for normalization of gene expression in human cancer research. BMC Bioinformatics 20, 245 (2019). https://doi-org/10.1186/s12859-019-2809-2
- qPCR makalesi: Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 2001;25(4):402-408. doi:10.1006/meth.2001.1262
- GeneCards: https://www.genecards.org/
- ExpressionAtlas: https://www.ebi.ac.uk/gxa/home
- Primer Verim Hesaplama: http://www.sigmaaldrich.com/technical-documents/protocols/biology/qpcr-efficiency-determination.html
- Örnek bir primer verim hesaplama grafiği: https://www.novusbio.com/products/afm-primer_nbp1-71653
